|

|

|

|
|

|
 > Tumorklassifizierung mit Hilfe der Genomik <
> Tumorklassifizierung mit Hilfe der Genomik <
|
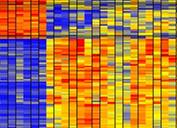
Genexpression von gesunden
Zellen und von Krebszellen
mehr Bild: G. Marra |
|
Der Projektleiter Giancarlo Marra der Arbeitsgruppe von Prof. Jiricny klassifiziert Dickdarmtumore mit Hilfe genomischer Analysen. Aufgrund der Klassierung sollen in Zukunft Empfehlungen zur Behandlung und Aussagen über den zu erwartenden Verlauf der Erkrankung gemacht werden können. Die Analysen werden mit DNA-Chips durchgeführt, die pro Chip DNA-Fragmente von rund 25'000 menschlichen Genen aufweisen. Früher musste man sich bei der Tumortypisierung auf einzelne Gene konzentrieren. Heute kann man mit Hilfe der Chips die Aktivität aller Gene in einem Tumor direkt bestimmen.
Für die Untersuchung wird Gewebe von frisch entfernten Tumoren verwendet. Dies ist wichtig, weil die RNA im Gewebe schnell abgebaut wird. Ca. 50 Milligramm Gewebe genügen für eine Analyse. Das Gewebe wird zerkleinert, die Zellen zerstört, die RNA mit Standardmethoden extrahiert, zuerst in komplementäre cDNA und dann in mit Fluoreszenzmarkern gekoppelte cRNA umgeschrieben. Die markierte cRNA wird auf einen DNA-Chip gebracht, wo sie mit den DNA-Fragmenten auf dem Chip hybridisiert. Die im Tumor aktiven Gene werden schliesslich auf dem Chip durch farbig leuchtende Punkte erkennbar. Je nach Farbe und Leuchtstärke ist die Genexpression im Tumor stärker oder schwächer als im normalen Gewebe. Das erhaltene Farbmuster ist für jedes Gewebe bzw. jeden Tumor charakteristisch. Die Muster werden in einen Computer eingelesen und mit Hilfe einer Software kann ein Tumor aufgrund seines Genexpressionsmusters klassifiziert werden.
Um eine hohe statistische Aussagekraft der Analyse zu erhalten, werden je 10 Gewebeproben des Tumors mit 10 Proben von gesundem Gewebe, d.h. mit gesunder Darmschleimhaut derselben Person, verglichen.
|
|
|
|



